特定的微生物特征或能增强仔猪对PRRSV-1的抵抗力
点击↖硕腾猪业关注我,加★星标★

温馨提示:由于微信公众号试行乱序推送,您可能不再能准时收到推送。为了不错过硕腾猪业消息,请将“硕腾猪业”公众号点亮为星标账号,并常点文末右下角的“在看”。
在早期感染高毒性PRRSV-1毒株后,那些存活下来的仔猪,在三周龄时相较于未能存活的仔猪,其鼻腔微生物群展现出更强的多样性。
反观未能存活的仔猪,其鼻腔微生物群则呈现出以大肠杆菌为主导的失调状态。这一现象表明,特定的微生物特征或许能够助力仔猪增强对PRRSV-1的抵抗力。
这些差异似乎并不只是局限于PRRSV感染,还与疫情暴发前各批次仔猪的存活情况有关。
材料和方法
该研究在一家1400头母猪的猪场进行,该猪场于2021年12月遭遇了一种高致病性PRRSV-1毒株的侵袭。研究人员利用在产房和保育舍定期监测呼吸道病原体时,对来自不同窝的31头三周龄PPRSV感染仔猪进行了细致检查。
待保育期结束,将这些仔猪区分为存活组(S)和非存活组(D)。通过 RT-qPCR技术确认了PRRS以及IAV(猪流感病毒)感染情况,同时对鼻拭子的DNA进行了16S rRNA测序。
后续运用QIIME2软件开展生物信息学分析,精准确定了微生物的多样性、丰富度以及分类学特征,并借助统计测试和多样性指标(例如Chao1和Shannon指数)对各组别进行了全面比较。此外,利用Songbird、ANCOM以及PICRUSt2工具分析了差异丰富的分类群和推测的功能途径。最终,在RStudio平台中完成了微生物组数据的整合与可视化工作。
结果与讨论
S组和D组猪仔的鼻腔微生物群组成存在显著差异。存活猪仔表现出更高的菌群丰富度(Chao1指数,P=0.001),但多样性相似(Shannon指数,P=0.66)。β多样性分析显示出S组和D组之间的明显聚类,微生物差异部分受母猪不同窝次影响。有趣的是,D组中肠杆菌科和大肠杆菌的丰度更高。
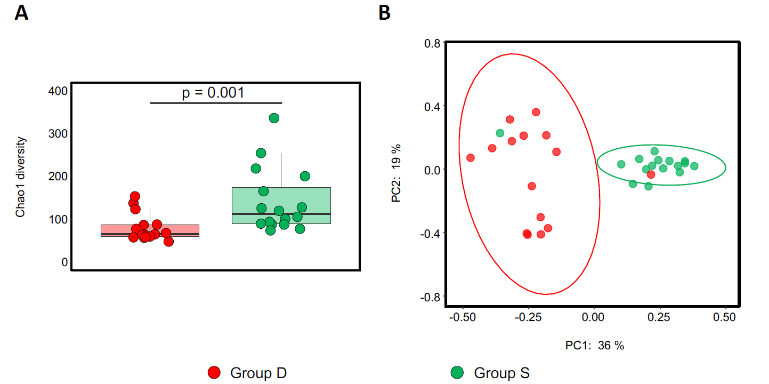
图1 使用chao1指数测量S(绿色)和D(红色)组的α多样性
疫情暴发前样本根据存活结果表现出类似的聚集情况,这表明微生物群差异早于PRRSV-1暴发,并且与整体存活率增加有关。母猪微生物群也因窝产存活率以及疫情暴发前后的时期存在差异,将微生物群组成跨代联系起来。
原文链接:https://umnswinenews.com/2025/02/21/nasal-microbial-diversity-is-associated-with-survival-in-piglets-infected-by-a-highly-virulent-prrsv%e2%80%911-strain/
点击下方小程序码,免费猪病问诊
往期内容回顾
点个在看再走吧~






{{item.content}}